Mehr als 2 Merkmale#
Es folgt ein Überblick über Methoden der deskriptiven Statistik für multivariate Daten, wenn mehr als 2 Merkmale gemessen/erhoben wurden. Zu beachten ist hier wieder welche Messbarkeit die erhobenen Merkmale haben.
Maßzahlen#
Vektor mit Maßzahlen#
Die Maßzahlen der univariaten Statistik können pro Merkmal erhoben werden und dann als Vektor oder Tabelle dargestellt werden. Liegen metrische Daten vor, so kann z.B. ein Mittelwertvektor gebildet werden. Dies ist zeigt natürlich nicht den Zusammenhang der Daten, sondern behandelt sie wie einzelne getrennt voneinander erhobene Stichproben.
Umsetzung in R
Hier bietet es sich an, den apply()-Befehl zu nutzen. Damit kann man eine Funktion auf die Spalten (oder Zeilen) eines data.frames anwenden.
apply(iris[,1:4], 2, mean)
- Sepal.Length
- 5.84333333333333
- Sepal.Width
- 3.05733333333333
- Petal.Length
- 3.758
- Petal.Width
- 1.19933333333333
Erkärung des Codes
iris[,1:4]wählt die ersten 4 Spalten vonirisaus. In der 5. Spalte stehen die Spezies, die möchten wir hier nicht betrachten.Die
2steht für Spalten, d.h. wir wollen die Funktion auf die Spalten anwenden. Eine1würde bedeuten, dass wir die Funktion auf die Zeilen anwenden wollen.meanheißt die Funktion die wir auf jede einzelne Spalte anwenden wollen
Es ist auch möglich eine Funktion selbst zu definieren und diese in den apply-Befehl einzusetzen. Die ist zum Beispiel nötig, wenn wir den Variationskoeffizient pro Spalte ausrechnen wollen.
varkoeff <- function(x){ sd(x)/mean(x)}
apply(iris[, 1:4], 2, varkoeff)
- Sepal.Length
- 0.14171125977944
- Sepal.Width
- 0.142564201353041
- Petal.Length
- 0.46974407484286
- Petal.Width
- 0.635551141434419
Erkärung des Codes
varkoeff <- function(x){ ... }bedeutet wir definieren eine neue Funktion. Sie sollvarkoeffheißen. Hier kann man sich einen beliebigen Namen ausdenken. Der Ausdruckfunction(x)sagt, dass es eine Funktion ist und das Argumentxheißt. In die geschweiften Klammern schreibt man, was die Funktion mit demxmacht, also die Bildungsvorschrift.sd(x)/mean(x)ist die Bildungsvorschrift in unserer Funktion: Berechne die Standardabweichung vonxund teile diesen Wert durch das arithmetische Mittel vonx.
Der summary-Befehl#
Eine schnelle Übersicht über viele Maßzahlen liefert der Befehl summary().
summary(iris)
Sepal.Length Sepal.Width Petal.Length Petal.Width
Min. :4.300 Min. :2.000 Min. :1.000 Min. :0.100
1st Qu.:5.100 1st Qu.:2.800 1st Qu.:1.600 1st Qu.:0.300
Median :5.800 Median :3.000 Median :4.350 Median :1.300
Mean :5.843 Mean :3.057 Mean :3.758 Mean :1.199
3rd Qu.:6.400 3rd Qu.:3.300 3rd Qu.:5.100 3rd Qu.:1.800
Max. :7.900 Max. :4.400 Max. :6.900 Max. :2.500
Species
setosa :50
versicolor:50
virginica :50
Kovarianzmatrix und Korrelationsmatrix#
Zeigt die Kovarianzen (oder Korrelationskoeffizienten) zwischen allen Variablenpaaren (Diagonalen = Varianzen). Um dies zu verstehen, wiederholen Sie gern Maßzahlen im Abschnitt Zwei metrische Merkmale.
An Stelle \(i,j\) der Matrix steht die Kovarianz (oder der Korrelationskoeffizient) vom \(i\)-tem und \(j\)-tem Merkmal. Die Kovarianz (bzw Korrelation nach Pearson) lässt sich natürlich nur zwischen stetigen Merkmalen erheben. Auf der Diagonalen der Kovarianzmatrix steht jeweils die Varianz. Auf der Diagonalen der Korrelationsmatrix steht immer eine \(1\), denn dies ist der Korrelationskoeffizient von eine Spalte mit sich selbst.
Merke:
Kovarianzmatrix:
An Stelle \((i,j)\) steht die Kovarianz von Spalte \(i\) und Spalte \(j\).
Die Kovarianzmatrix ist symmetrisch: An Stelle \((i,j)\) steht immer der gleiche Wert wie an Stelle \((j,i)\).
Auf der Diagonalen stehen die Varianzen: An Stelle \((i,i)\) steht die Varianz der Daten in Spalte \(i\).
Korrelationsmatrix:
An Stelle \((i,j)\) steht der Korrelationskoeffizient von Spalte \(i\) und Spalte \(j\).
Die Korrelationsmatrix ist symmetrisch: An Stelle \((i,j)\) steht immer der gleiche Wert wie an Stelle \((j,i)\).
Auf der Diagonalen stehen immer \(1\)en.
Umsetzung in R
Wendet man die (schon bekannten) Befehle cov und cor auf einen data.frame mit metrischen Daten an, so wird die Korrelationsmatrix bzw die Kovarianzmatrix erstellt.
cov(iris[,1:4])
cor(iris[,1:4])
| Sepal.Length | Sepal.Width | Petal.Length | Petal.Width | |
|---|---|---|---|---|
| Sepal.Length | 0.6856935 | -0.0424340 | 1.2743154 | 0.5162707 |
| Sepal.Width | -0.0424340 | 0.1899794 | -0.3296564 | -0.1216394 |
| Petal.Length | 1.2743154 | -0.3296564 | 3.1162779 | 1.2956094 |
| Petal.Width | 0.5162707 | -0.1216394 | 1.2956094 | 0.5810063 |
| Sepal.Length | Sepal.Width | Petal.Length | Petal.Width | |
|---|---|---|---|---|
| Sepal.Length | 1.0000000 | -0.1175698 | 0.8717538 | 0.8179411 |
| Sepal.Width | -0.1175698 | 1.0000000 | -0.4284401 | -0.3661259 |
| Petal.Length | 0.8717538 | -0.4284401 | 1.0000000 | 0.9628654 |
| Petal.Width | 0.8179411 | -0.3661259 | 0.9628654 | 1.0000000 |
Graphische Darstellungen#
Heatmap#
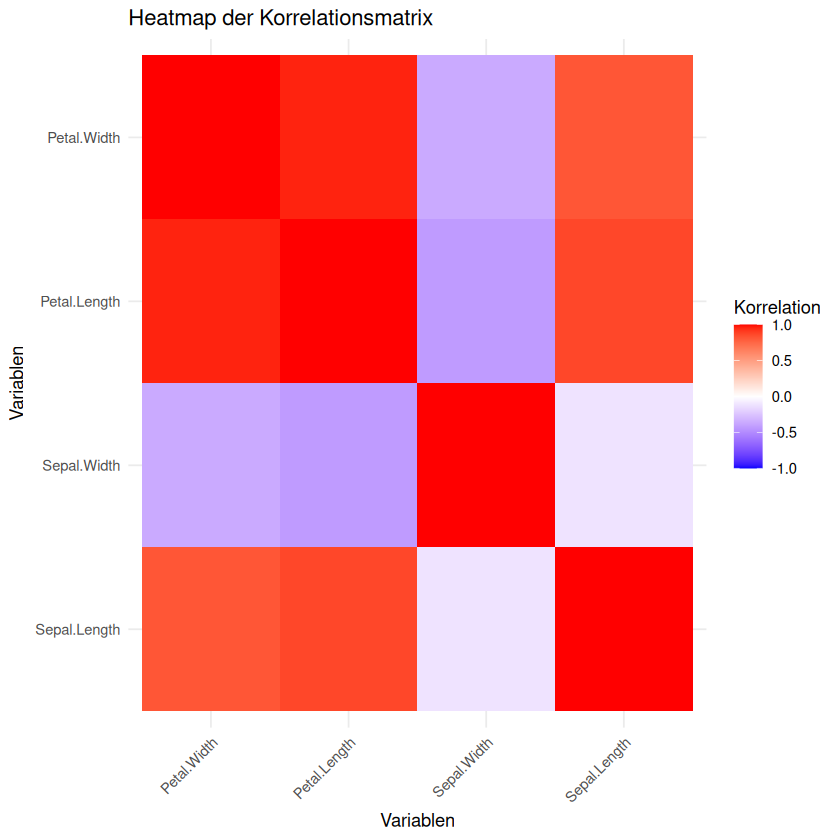
Eine Heatmap ist eine Art von visueller Darstellung, die verwendet wird, um komplexe Daten zu visualisieren und zu analysieren. Sie zeigt die Beziehungen zwischen verschiedenen Variablen oder Werten in einer Tabelle oder Matrix.
Eine Heatmap besteht aus einer Tabelle oder Matrix, in der jede Zelle einen Wert darstellt. Die Werte werden in einer Farbskala dargestellt, wobei die Helligkeit oder Intensität der Farbe die Größe oder Stärke des Wertes anzeigt. Die Farben können auch unterschiedliche Nuancen haben, um die Art der Beziehung zwischen den Variablen anzuzeigen.
Eine Heatmap für eine Korrelationsmatrix ist eine besondere Art von Heatmap, die verwendet wird, um die Korrelationen zwischen verschiedenen Variablen in einer Datenmenge zu visualisieren. Hier werden die Korrelationen zwischen den Variablen in einer Farbskala dargestellt. Dadurch kann man schnell erkennen, welche Variablen stark miteinander korreliert sind und welche nicht.
Umsetzung in R: Varinate 1 ohne zusätzliches Paket
# Korrelationsmatrix berechnen
korrelationsmatrix <- cor(iris[, 1:4])
# Heatmap zeichnen
heatmap(korrelationsmatrix, # korrelationsmatrix ist Input
symm=TRUE, # sagt, dass symm. Matrix eigegeben wird
revC = TRUE, # sort für die richtige Reihenfolge d. Zeilen
Rowv = NA,Colv = NA,) # kein Dendrogramm zeichnen
Umsetzung in R: Varinate 2 mit reshape2 und ggplot2
Mit ggplot2 sieht der Heatplot etwas moderner aus. Außerdem haben wir die Möglichkeit die eine Legende für die Farbscala hinzuzufügen.
library(ggplot2)
library(reshape2)
# Korrelationsmatrix berechnen
korrelationsmatrix <- cor(iris[, 1:4])
# Umstrukturieren der Matrix für ggplot2
korrelationsmatrix_melted <- melt(korrelationsmatrix)
ggplot(korrelationsmatrix_melted,
aes(factor(Var1, levels = rev(levels(Var1))), Var2, fill = value)) +
geom_tile() +
scale_fill_gradient2(low = "blue", high = "red", mid = "white", midpoint = 0, limit = c(-1, 1), space = "Lab", name = "Korrelation") +
theme_minimal() +
theme(axis.text.x = element_text(angle = 45, vjust = 1, hjust = 1)) +
labs(title = "Heatmap der Korrelationsmatrix", x = "Variablen", y = "Variablen")
Erläuterung des Codes
library(reshape2): Hier laden wir die Bibliothekreshape2, die wir später für die Umstrukturierung der Korrelationsmatrix benötigen.korrelationsmatrix <- cor(iris[, 1:4]): Berechnet die Korrelationsmatrixkorrelationsmatrix_melted <- melt(korrelationsmatrix): Umstrukturierung der Matrix, so dass sie als Eingabe fürggplot2passt. Diemelt()-Funktion erstellt eine neue Tabelle, in der jede Zeile eine einzelne Zelle der ursprünglichen Matrix darstellt. Die neuen SpaltenVar1undVar2enthalten die Namen der Variablen, die korreliert sind, und die Spaltevalueenthält die Korrelation zwischen den Variablen.ggplot(korrelationsmatrix_melted, ...)der plot Befehl vonggplot2aes()definiert die Ästhetik der Heatmap.Die
factor()-Funktion wird verwendet, um die Variablen in der Var1-Spalte in der richtigen Reihenfolge anzuzeigen.Die
geom_tile()-Funktion erstellt die Heatmap selbst.Die
scale_fill_gradient2()-Funktion definiert die Farbskala für die Heatmap.Die
theme_minimal()-Funktion passt die Optik leicht an (ist optional)Die
theme()-Funktion setzt die Anzeige der Variablen in derVar1-Spalte.Die
labs()-Funktion setzt den Titel und die Beschriftungen für die Heatmap.
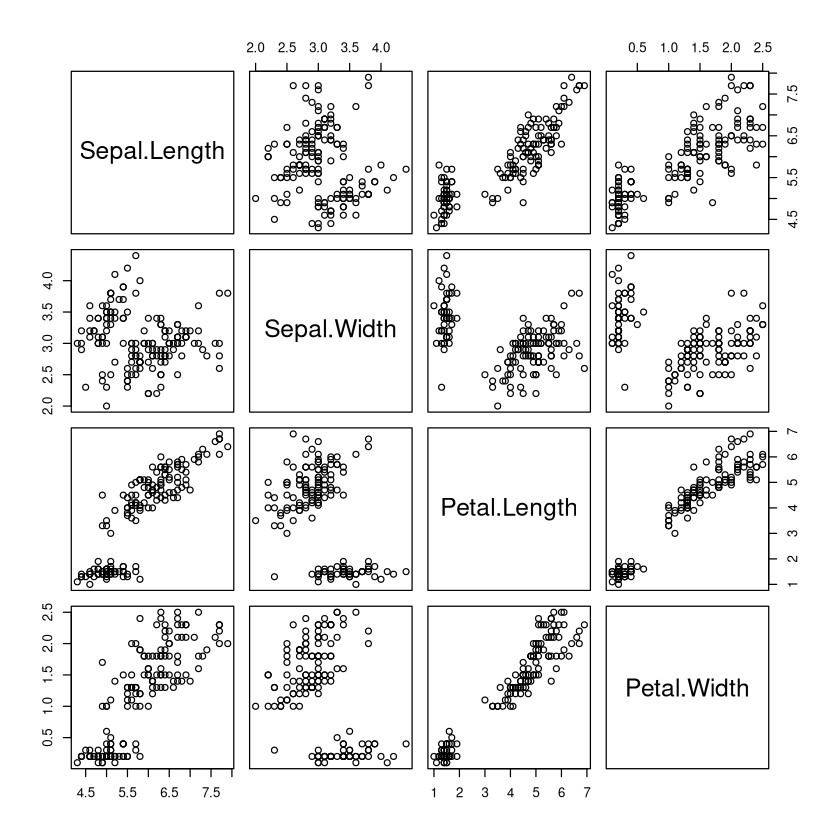
Streudiagramm-Matrix#
Die paarweise Darstellung aller Streudiagramme der möglichen Variablenkombinationen.
Dies ist schnell erreicht mit dem Befehl pairs().
3D-Streudiagramme#
Für drei Variablen lässt sich noch ein Streudiagramm zeichnen. Die ist auch möglich mit Interaktion/Rotation. Halten Sie dazu in der Grafik die linke Maustaste gedrückt und bewegen Sie den Curser.
Wir plotten zunächst 3 Merkmale aus dem iris- Datensatz. Die Färbung wird entsprechend der Spezies vorgenommen.
library(plotly) # Paket für 3d-Plots
Show code cell output
Attache Paket: ‘plotly’
Das folgende Objekt ist maskiert ‘package:ggplot2’:
last_plot
Das folgende Objekt ist maskiert ‘package:stats’:
filter
Das folgende Objekt ist maskiert ‘package:graphics’:
layout
plot_ly(data = iris , # plot_ly ist der Plot-Befehl aus "plotly",
x = ~Sepal.Length, # data= legt Datensatz fest
y = ~Sepal.Width, # x,y,z = ordnet den Achsen die Spalte zu
z = ~Petal.Width, #
color = ~Species, # Farbe entsprechend der Spezies
type="scatter3d", # es soll ein 3d-Plot entstehen
mode="markers", # werte sollen als Punkte dargestellt werden
size = 0.2) # Größe der Punkte
# Ein weiteres Beispiel mit zufälligen Daten
set.seed(123) # damit bleibt der Zufall jedes mal gleich
temp <- rnorm(100, mean=30, sd=5) # zufällige Temperaturen auswürfeln
pressure <- rnorm(100) # zufälligen Druck auswürfeln
dtime <- 1:100 # Zeitpunkte zuordnen
plot_ly(x=temp, y=pressure, z=dtime,
type="scatter3d", mode="markers",
color=temp) # Farbe entsprechend der Temperatur
Parallele Koordinaten#
Was ist ein Parallel Coordinate Plot?
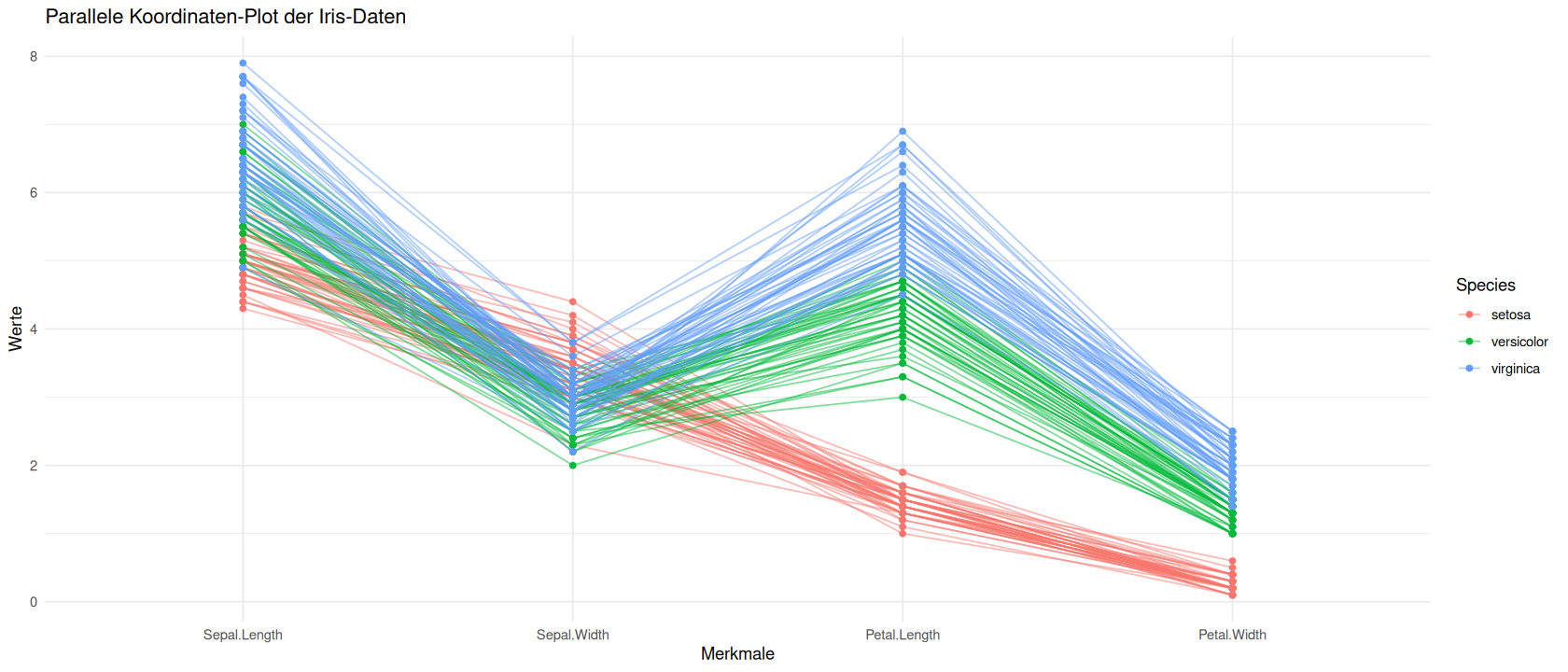
Ein Parallel Coordinate Plot (deutsch: Parallele Koordinaten-Diagramm) ist eine Visualisierungsmethode zur Darstellung mehrerer numerischer Variablen gleichzeitig. Dabei wird jede Variable auf einer eigenen vertikalen Achse dargestellt, und jede Beobachtung (Zeile im Datensatz) wird als Linie gezeichnet, die die Werte der Beobachtung über die Achsen hinweg verbindet.
Diese Diagrammart eignet sich besonders zur Darstellung von multivariaten Daten mit mehreren numerischen Variablen. Alle Variablen werden auf parallelen Achsen gezeigt, um Muster in Beobachtungen zu erkennen.
Wie erstellt man einen Parallel Coordinate Plot in R?
Für die Erstellung von Parallel Coordinate Plots in R wird häufig das Paket GGally verwendet, das eine Erweiterung von ggplot2 ist. Wir zeigen dies am Beispiel des iris Datensatz.
library(ggplot2) # Pakte um schöne Grafiken zu erstellen
library(GGally) # Paket um den Parallel-Koordianten-Plot zu erstellen
Registered S3 method overwritten by 'GGally':
method from
+.gg ggplot2
options_orig<-options() # Anzeigeoptionen speichern
options(repr.plot.width = 14, repr.plot.height = 6) # Anzeigeoptionen ändern (Breite Grafik)
# Parallel Coordinate Plot
ggparcoord(data = iris,
columns = 1:4, # Die Spalten 1 bis 4 (numerische Variablen)
groupColumn = 5, # Gruppierung nach Species
scale = "globalminmax", # Werte auf gleiche Skala bringen
alphaLines = 0.5, # Transparenz der Linien
showPoints = TRUE) + # Punkte für einzelne Werte anzeigen
labs(title = "Parallele Koordinaten-Plot der Iris-Daten",
x = "Merkmale", # Titel und Achsen
y = "Werte") + # Bezeichnungen
theme_minimal() # optionale optische Anpassung
options(options_orig) # Anzeigeoptionen zurücksetzen
Interpretation
Beim Interpretieren eines Parallel Coordinate Plots achtet man auf folgende Aspekte:
Gruppenunterschiede: Linien von verschiedenen Gruppen (z.B. Arten) haben oft ähnliche Verläufe.
Korrelationen: Wenn zwei benachbarte Achsen fast parallele Linien zeigen, deutet dies auf eine positive Korrelation hin. Kreuzen sich die Linien häufig, könnte eine negative Korrelation vorliegen.
Ausreißer: Einzelne Linien, die sich stark von den anderen unterscheiden, können auf Ausreißer hinweisen.
Streuung: Eine breite Streuung der Linien zeigt eine hohe Varianz in den Daten.
Vor- und Nachteile
Vorteile |
Nachteile |
|---|---|
Darstellung vieler Variablen |
Schwer lesbar bei großen Datenmengen |
Vergleich von Gruppen möglich |
Interpretation kann schwierig sein |
Identifikation von Ausreißern möglich |
Linien können sich stark überlagern |
Visualisierung von Korrelationen |
Reihenfolge der Achsen beeinflusst das Ergebnis |